- Biographie
- Travaux
- Contact
Les informations ci-après se réfèrent à la date de la remise du Prix.
Citoyen suisse, né en 1940, Ulrich K. LAEMMLI est professeur dans les départements de biochimie et de biologie moléculaire de la Faculté des Sciences de l’Université de Genève.
Ulrich K. LAEMMLI est responsable de nombreuses découvertes dans le domaine de la structure des chromosomes. Grâce à une véritable dissection biochimique, il a montré que cette structure repose sur un « échafaudage » de protéines non-histoniques, auquel le matériel génétique s’attache en des points précis appelés SAR (scaffold associated regions). Une série d’explorations biochimiques très originales lui ont permis de mettre en lumière les équilibres dynamiques dont cet échafaudage est le siège. Ces découvertes ont renouvelé profondément notre vision de la structure des chromosomes. Celle-ci repose sur une organisation en domaines fonctionnels distincts, à la fois très compacts mais néanmoins suffisamment flexibles pour permettre les interactions dynamiques dans lesquelles l’ADN est engagé, comme par exemple l’assemblage des chromosomes après la division cellulaire et l’expression sélective des gènes.
Les travaux d’Ulrich K. LAEMMLI visent désormais à créer des molécules capables d’interagir de manière ciblée sur la structure des chromosomes, ce qui aurait des retombées importantes en chimiothérapie anticancéreuse.
Le Prix Louis-Jeantet de médecine permettra à Ulrich K. LAEMMLI d’acquérir des équipements d’analyse et de synthèse de peptides, et de s’adjoindre un collaborateur supplémentaire pour la poursuite de ce projet.
Travaux de recherche
Comprendre la structure des chromosomes a été dès l’origine un objectif majeur de la biologie moléculaire. En effet, les 46 chromosomes que contient chacune de nos cellules constituent la » bibliothèque » génétique que nous avons héritée de nos parents. Cette bibliothèque génétique est présente en double exemplaire puisqu’un jeu de 23 chromosomes nous vient de notre mère, et l’autre de notre père. Le constituant matériel de notre patrimoine génétique, l’ADN, est empaqueté à l’intérieur des chromosomes du fait de son association avec certaines protéines.
L’ADN du génome haploïde (l’ADN présent dans un jeu de 23 chromosomes) comprend plus de trois milliards de bases, c’est-à-dire que trois milliards de lettres A, C, G ou T sont nécessaires à l’écriture de l’information génétique contenue dans ces 23 chromosomes. Si l’on pouvait déployer cet ADN et placer bout à bout les doubles hélices débarrassées de leur gangue protéique, on obtiendrait une longueur d’un mètre d’ADN. Or dans une cellule humaine, cet ADN est emballé dans un volume extrêmement réduit, d’où l’un des paradoxes de la biologie moléculaire : comment l’ADN peut-il être comprimé dans une structure très compacte sans affecter néanmoins le caractère accessible de l’information qu’il porte, ni empêcher sa copie fidèle, lettre par lettre, à chaque division cellulaire ? En effet, dans une cellule donnée, des dizaines de milliers de gènes sont actifs et exprimés et cette expression génétique est rigoureusement contrôlée suivant les étapes de la différenciation cellulaire. De plus, lors de la division cellulaire, l’ensemble du matériel génétique est fidèlement copié pour donner naissance à deux génomes identiques, ce qui implique également un » déballage » étroitement contrôlé de l’ADN des chromosomes, suivi d’une reconstruction de la structure chromosomique.
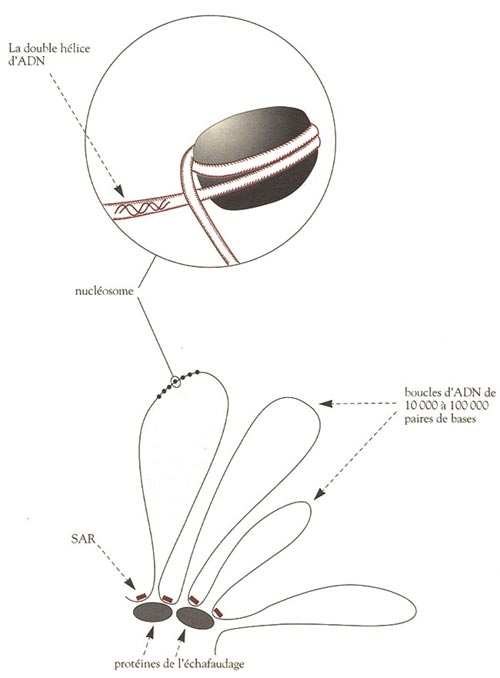
La molécule linéaire d’ADN n’est donc pas repliée au hasard comme dans une pelote de laine par exemple. Elle est organisée en une structure bien précise à plusieurs niveaux hiérarchisés. Le premier niveau est une structure appelée nucléosome et correspond à un enroulement de l’ADN autour de plusieurs molécules de protéines, les histones. L’enchaînement des nucléosomes, souvent comparé aux perles d’un collier, est lui-même pris dans un enroulement d’ordre supérieur qui est maintenu par un » échafaudage » de protéines différentes des histones. Cet échafaudage est l’élément structurel le plus important du chromosome. Il a été découvert par Ulrich Laemmli, qui a montré que si l’on débarrasse les chromosomes de leurs histones par divers traitements chimiques, les chromosomes conservent néanmoins leur forme générale et leur structure d’ensemble. Cela démontre l’existence d’un assemblage structural distinct, qui ne dépend pas des histones (ni des nucléosomes).
Cet échafaudage (scaffold) résulte de l’interaction entre certaines régions de l’ADN appelées SAR (scaffold-associated regions) et de certaines protéines dont la plus importante est la topoisomérase II. Cette interaction est à l’origine de l’échafaudage et délimite du même coup des boucles d’ADN de plusieurs dizaines de milliers de bases, situées entre régions SAR successives. Ces boucles sont très importantes car elles constituent en fait des domaines fonctionnels du génome, qui peuvent être sujets à des régulations biologiques indépendantes. En effet, il est probable que les SAR se situent entre les gènes et non à l’intérieur de ceux-ci : ces régions – ainsi que la structure en échafaudage qui en résulte – délimitent donc des » chapitres » de l’information génétique accessibles indépendamment les uns des autres. De plus, il est significatif que la topoisomérase II intervienne dans la mise en place de ces structures, car il s’agit en fait d’une enzyme capable de défaire les » noeuds » dans la molécule d’ADN. En effet, lors de la réplication de l’ADN, la double hélice s’entrouvre pour permettre la synthèse de nouveaux brins d’ADN, et en l’absence de topoisomérase II le processus de réplication serait très vite bloqué par la formation d’un fouillis de chaînes emmêlées.
L’échafaudage et les régions SAR jouent donc un rôle crucial dans le maintien de l’intégrité structurale et fonctionnelle des chromosomes. Ulrich Laemmli explore désormais les implications médicales potentielles de ces découvertes, dans le domaine de la chimiothérapie anticancéreuse. Comme le trait distinctif des cellules tumorales est qu’elles n’arrêtent pas de se diviser, certains médicaments anti-tumoraux sont conçus comme des poisons de la réplication de l’ADN. Or une molécule qui s’attacherait de manière spécifique aux régions SAR pourrait empêcher l’assemblage des chromosomes de la cellule tumorale. Ce ciblage spécifique des régions SAR représente un grand progrès par rapport à des molécules qui réagissent avec l’ADN de façon non spécifique, car il permet d’envisager un effet biologique avec des doses faibles et donc une toxicité moindre.
Prof. Ulrich Laemmli
Département de biologie moléculaire
Faculté des sciences
SCIENCES II
Quai Ernest-Ansermet, 30
CH – 2111 Genève 4
Tél.: +41 22 702 61 22
Fax: +41 22 329 06 96
E-mail: Ulrich.Laemmli@molbio.unige.ch
Site Internet:
Université de Genève, Département de biologie moléculaire
