- Biographie
- Travaux
- Contact
Les informations ci-après se réfèrent à la date de la remise du Prix.
Wolfgang BAUMEISTER est le directeur de l’Institut Max-Planck de Biochimie à Martinsried près de Munich, dont il dirige également le Département de Biologie structurale. Wolfgang BAUMEISTER est citoyen allemand. Il est né en 1946.
L’architecture de la cellule comporte un grand nombre de membranes et de complexes de protéines qu’il était impossible, jusqu’à récemment, de visualiser en trois dimensions sous haute résolution. Les scientifiques soit prenaient des images en deux dimensions de coupes fines par microscopie électronique, soit désintégraient les cellules pour isoler leurs constituants et les analyser individuellement. Tout ceci a considérablement changé avec les travaux de Wolfgang BAUMEISTER et de ses collaborateurs qui ont affiné une technique appelée tomographie par cryomicroscopie électronique. Cette méthode utilise aussi le microscope électronique, mais analyse des objets mis rapidement en congélation, ce qui préserve leur structure. Wolfgang BAUMEISTER a introduit un dispositif qui permet de prendre simultanément de nombreuses images sous différents angles, qui sont ensuite assemblées par ordinateur pour former des images en trois dimensions. Wolfgang BAUMEISTER a d’abord apporté une contribution essentielle à l’étude de la structure et de la fonction du protéasome, un grand complexe de protéines nécessaire à la dégradation intracellulaire des protéines. Récemment, il a montré que la tomographie par cryomicroscopie électronique permettait de visualiser de tels complexes dans leur environnement fonctionnel : la cellule intacte.
Avec le Prix Louis-Jeantet de médecine, Wolfgang BAUMEISTER se propose d’améliorer encore la résolution déjà élevée de la tomographie par cryomicroscopie électronique et d’étudier l’architecture tridimensionnelle des ensembles moléculaires qui constituent les synapses des cellules nerveuses. On pourra ainsi avoir une idée des changements qui interviennent au cours de l’activation synaptique. Il prévoit d’agrandir son groupe avec l’engagement d’un nouveau collaborateur spécialisé.
Notice biographique
De nationalité allemande, Wolfgang Baumeister est né en 1946. Il a étudié la biologie aux Universités de Münster et de Bonn. En 1973, il obtient son doctorat au Département de biophysique de l’Université de Düsseldorf. Il resta dans ce département en qualité de collaborateur scientifique jusqu’en 1981, où il se rendit pour un séjour de deux ans au Laboratoire de Cavendish à l’Université de Cambridge (Grande-Bretagne). En 1983, il devient chef de groupe à l’Institut Max-Planck de Biochimie à Martinsried près de Munich. En 1984, il est nommé professeur à l’Université de Düsseldorf et, en 1987, professeur à l’Université Technique de Munich. Depuis 1988, il est le Directeur de l’Institut Max-Planck de Biochimie à Martinsried près de Munich, où il dirige également le Département de biologie structurale. Wolfgang Baumeister a reçu plusieurs autres distinctions: le Prix Ernst-Ruska en 1982, la médaille Otto-Warburg en 1998, et, en 2001, le Prix de la Fondation Feldberg en Angleterre, le Prix de la Fondation scientifique Fourmentin-Guilbert en France et le Prix Karl Heinz Beckurts. Depuis 2000, Wolfgang Baumeister est professeur honoraire de l’Université Technique de Munich. Il est également membre de l’EMBO (depuis 1988), membre de l’Académie Allemande des Sciences Naturelles Leopoldina (depuis 2001) et membre honoraire étranger de l’Académie Américaine des Arts et des Sciences (depuis 2002).
Tomographie électronique
La tomographie par microscopie électronique n’est en aucun cas une technique nouvelle d’imagerie, mais elle a récemment pris un essor particulier. L’arrivée de microscopes électroniques pilotés par ordinateur et l’automatisation des processus d’acquisition d’images détaillées ont permis d’obtenir des tomogrammes à résolution moléculaire de structures aussi grandes et complexes que des cellules procaryotes ou eucaryotes intactes enrobées d’une fine couche de glace amorphe. L’imagerie non invasive de cellules entières vitrifiées est probablement le domaine où l’impact de la tomographie sera le plus grand. La cellule apparaît comme un assemblage supramoléculaire géant. Cependant, à l’échelle nanométrique, le cytoplasme est en général un territoire inexploré. De la même manière que l’on obtient des informations précieuses sur le fonctionnement de macromolécules par l’observation à haute résolution de leurs structures tridimensionnelles, on parviendra à une meilleure compréhension des fonctions cellulaires grâce à la possibilité de visualiser les macromolécules dans un contexte cellulaire non perturbé.
Les tomogrammes de cellules à une résolution moléculaire sont essentiellement des images en trois dimensions du protéome entier de la cellule. Ils fournissent des informations sur l’interactome, autrement dit sur la distribution et les interactions des macromolécules dans le cytoplasme. L’exploitation de ces informations se heurte cependant à deux problèmes majeurs : les cryotomogrammes sont » contaminés » par un important » bruit de fond » et déformés par des données incomplètes obtenues lors de la récolte des images. En outre, le cytoplasme est » surpeuplé » et les molécules qui s’y trouvent se touchent littéralement les unes les autres. Dans ces conditions il est généralement impossible d’interpréter les tomogrammes par une simple lecture, sauf pour les formes facilement reconnaissables comme les membranes ou le cytosquelette.
On peut toutefois utiliser l’une ou l’autre des techniques de reconnaissance de formes pour détecter et identifier des molécules particulières. En effet, pour autant qu’il existe une structure en résolution haute ou moyenne de la molécule étudiée, on peut utiliser celle-ci comme structure de référence pour effectuer une recherche systématique de structures concordantes (voir la figure). Une telle approche par » signature moléculaire « , bien qu’exigeante au niveau informatique, peut facilement être rendue efficace par une recherche parallélisée. Dès que l’on a déterminé les coordonnées spatiales d’un complexe de protéines dans une cellule, on peut avoir recours à des sous-tomogrammes pour analyser plus en détail ce complexe et son voisinage. On peut utiliser l’analyse statistique à plusieurs variables de tels sous-tomogrammes pour observer des changements dans l’environnement fonctionnel d’une molécule.
On a pu montrer la faisabilité de cette approche en utilisant des » cellules fantômes « , c’est-à-dire des vésicules lipidiques remplies de macromolécules, qui offrent un scénario expérimental réaliste et permettent d’évaluer la précision de la méthode. Avec la résolution actuelle (non isotrope) de 4 à 5 nm, on ne peut détecter et visualiser que de grands complexes de protéines (> 400 kDa) dans un contexte cellulaire. Pour élargir le champ d’application de la tomographie cellulaire, il sera nécessaire d’améliorer sa résolution. Des considérations théoriques et l’amélioration continue des appareils (utilisation de la température de l’hélium liquide au lieu de celle de l’azote liquide, amélioration des détecteurs, possibilité d’inclinaison sur deux axes) font qu’une résolution au voisinage de 2 nm n’est pas un objectif irréaliste.
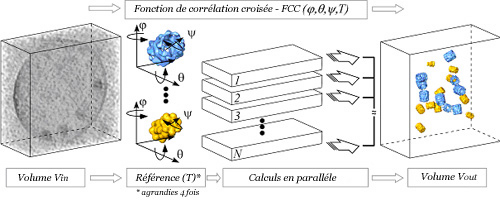
Figure 1 : Cartographie de la distribution spatiale de complexes de protéines et de leurs interactions au sein d’une cellule.
Un tomogramme à résolution moléculaire d’une cellule (Vin) est essentiellement une image en trois dimensions de l’ensemble du protéome de la cellule. Le » bruit de fond » et la » surpopulation » moléculaire gênent toutefois l’interprétation visuelle de l’information. Il faut donc utiliser la reconnaissance tridimensionnelle des formes pour extraire cette information. Une façon de faire efficace consiste à comparer l’information obtenue à des structures de référence. Il s’agit d’abord d’élucider la structure de référence de chaque complexe macromoléculaire à étudier (voire de fragments de ceux-ci), au moyen des diverses techniques susmentionnées, comme la cryomicroscopie électronique, la cristallographie au rayon X et la RMN, ou à l’aide de méthodes combinant ces techniques. On utilise ensuite ces structures de référence (T) pour rechercher les structures concordantes par corrélation croisée et on affine le résultat par une analyse statistique à plusieurs variables. Comme on ne connaît pas au départ ni les positions des complexes, ni leurs orientations, il est nécessaire de balayer Vin pour toutes les orientations possibles de chacune des structures de référence. Le résultat (Vout) montre les positions et les orientations des complexes dans la cellule. En principe, il devrait être possible de cartographier l’interactome cellulaire, autrement dit les relations et les interactions spatiales des principaux complexes de protéines d’une cellule.
Professeur Wolfgang BAUMEISTER
Directeur
Département de biologie structurale
Institut Max-Planck de Biochimie
Am Klopferspitz 18a
D – 82152 Martinsried b. München
Tél.: +4989 8578 2652/2642
Fax: +4989 8578 2641
E-mail: baumeist@biochem.mpg.de
Site Internet :
www.biochem.mpg.de/baumeister
